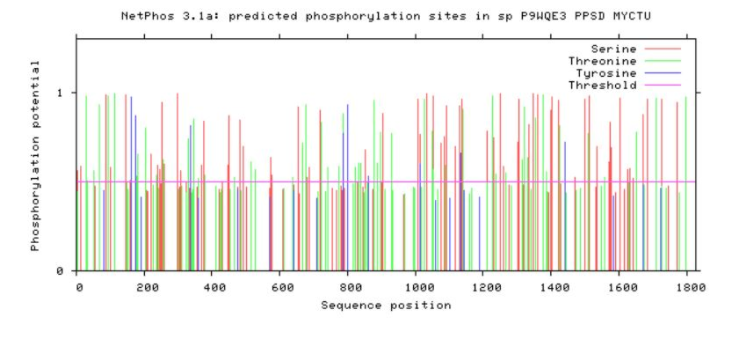
Mycobacterium tuberculosis

As bactérias do género Mycobacterium são bacilos (pleomórficos) gram positivo retos ou encurvados, cocobacilos, filamentosos ou formas ramificadas. Este organismo tem um comprimento de aproximadamente 1 a 10 μm, uma largura entre 0.2 e 0.6 μm e não formam esporo, flagelo ou cápsula. Apresenta uma parede complexa quanto à sua composição química, Lipídios de natureza hidrofóbica para a parede e ainda Bacilos Álcool Ácidos Resistentes (BAAR). Estas bactérias são intracelulares que infetam e proliferam-se no interior de macrófagos e linfócitos.
Sintomas
Os sintomas da tuberculose estão principalmente relacionados com o desenvolvimento da bactéria nos pulmões. Causando:
- Tosse seca e persistente com ou sem sangue
- Perda de peso
- Dor no peito ao tossir
- Dificuldade respiratória
No entanto, quando a bactéria se desenvolve em outros órgãos, é possível que surjam outros sintomas tais como:
- Cansaço excessivo
- Suor noturno
- Febre e inchaço no local que a bactéria está “instalada”
Tuberculose
A Tuberculose é a responsável pela morte de mais de 1 bilião de pessoas nos últimos 200 anos, esta doença cronica infeciosa é provocada pela bactéria Mycobacterium tuberculosis. Esta bactéria propaga-se pelo ar, quando uma pessoa infetada tosse, espirra, fala ou expele saliva podendo ser inalada por outro individuo. O sucesso da infeção e do desenvolvimento de tuberculose depende da sucessão de 4 passos: a fagocitose do bacilo, a multiplicação multicelular, o período de latência da infeção e da infeção ativa do pulmão.
Atualmente a Tuberculose continua a ser responsável pelo maior número de mortes provocadas por um único agente infecioso, causando cerca de 10,4 milhões de novos casos e 1,7 milhão de mortes por ano.
Ciclo de vida da bactéria
A resposta imunológica para a Mycobacterium tuberculosis não é completamente eficaz. Esta leva a bactéria para um estado latente, mas raramente a elimina. Infelizmente o estado latente da Mycobacterium tuberculosis é reversível, e a reativação da tuberculose é a fonte da maioria das transmissões (7). A infeção da tuberculose acontece em 4 etapas: A resposta inicial dos macrófagos, a etapa de crescimento, a etapa de controlo imunológico e a etapa do abcesso pulmonar. Estas quatro etapas ocorrem em cerca de 1 mês.